Биоинформатика
Молекулярная эволюция: как киты уходили под воду
Природа. 2016. №10. С.39–50.
Гельфанд М.С.
Биоинформатика
Молекулярная эволюция: как киты уходили под воду
Гипотеза о родстве китов и копытных была предложена больше ста лет назад, а сегодня уже доказано, что ближайшие родственники китов из современных животных — гиппопотамы. Какие данные об этом свидетельствуют и как события, происходившие на молекулярном уровне, соотносились с макроскопическими изменениями в анатомии и физиологии китов?
 |
|
По-видимому, первым родство китов и копытных предположил британский зоолог Фрэнк Беддард, известный прежде всего работами о червях, но написавший также в 1890 г. «Книгу о китах» [1]Beddard F.E. A book of whales. N.Y., 1900. Спустя столетие, к середине 1990-х годов, в среде зоологов и палеонтологов сложился консенсус: ближайшими родственниками или даже предками китов (Cetacea) стали считать вымерших хищных (!) копытных — мезонихий (Mesonychia). Однако таксономические взаимоотношения между ними и отрядами парнокопытных (Artiodactyla) и непарнокопытных (Perissodactyla) оставались не вполне ясными: то ли мезонихии — предки китов и парнокопытных, то ли парнокопытные — сестринская группа по отношению к китам и мезонихиям [2]O’Leary M.A., Geisler J.H. The position of Cetacea within mammalia: phylogenetic analysis of morphological data from extinct and extant taxa // Syst. Biol. 1999. V.48. P.455–490.[3]Arnason U., Gullberg A., Janke A. Mitogenomic analyses provide new insights into cetacean origin and evolution // Gene. 2004. V.333. P.27–34.Doi:10.1016/j.gene.2004.02.010 В любом случае все сходились во мнении, что ближайшая к китам группа из ныне живущих — парнокопытные и что вместе они образуют группу (кладу) китопарнокопытных (Cetartiodactyla). Молекулярные данные позволили существенно уточнить эту картину.
Филогенетические деревья
На основании анализа митохондриальных геномов была выдвинута гипотеза, согласно которой киты близки к подотряду жвачных парнокопытных (Ruminantia). Более далекими родственниками тех и других оказались остальные парнокопытные из подотрядов свинообразных (Suiformes), к которым тогда причисляли и гиппопотамов, и мозоленогих (Tylopoda), к которым относятся только верблюдовые [4]Graur D., Higgins D.G. Molecular evidence for the inclusion of cetaceans within the order Artiodactyla // Mol. Biol. Evol. 1994. V.11. P.357–364. Кроме того, результаты анализа белковых последовательностей цитохрома b [5]Irwin D.M., Arnason U. Cytochrome b gene of marine mammals: phylogeny and evolution // J. Mammal. Evol. 1994. V.2. P.37–55.Doi:10.1007/BF01464349 α- и β-казеина [6]Gatesy J., Hayashi C., Cronin M.A. et al. Evidence from milk casein genes that cetaceans are close relatives of hippopotamid artiodactyls // Mol. Biol. Evol. 1996. V.13. P.954–963. γ-фибриногена [7]Gatesy J. More DNA support for a Cetacea/Hippopotamidae clade: the blood-clotting protein gene gamma-fibrinogen // Mol. Biol. Evol. 1997. V.14. P.537–543. ретиноид-связывающего белка IRBP и фактора Виллебранда [8]Gatesy J., Milinkovitch M., Waddell V. et al. Stability of cladistic relationships between Cetacea and higher-level artiodactyl taxa // Syst. Biol. 1999. V.48. P.6–20. панкреатической рибонуклеазы [9]Kleineidam R.G., Pesole G., Breukelman H.J. et al. Inclusion of cetaceans within the order Artiodactyla based on phylogenetic analysis of pancreatic ribonuclease genes // J. Mol. Evol. 1999. V.48. P.360–368. указывали на то, что ближайшие родственники китов — гиппопотамы.
Однако существовали и опровергающие аргументы. В частности, писали о том, что формирование ветвей Ruminantia + Cetacea (в отличие от принятых до этого Ruminantia + Suiformes) и тем более Hippopotamidae + Cetacea зависит от параметров эволюционной модели, принятой при построении филогенетического дерева [10]Hasegawa M., Adachi J. Phylogenetic position of cetaceans relative to artiodactyls: reanalysis of mitochondrial and nuclear sequences // Mol. Biol. Evol. 1996. V.13. P.710–717. Аналогичным образом взаимоотношения между китами и подотрядами парнокопытных не могли быть определены при анализе митохондриальных 12S РНК [11]Montgelard C., Catzeflis F.M., Douzery E. Phylogenetic relationships of artiodactyls and cetaceans as deduced from the comparison of cytochrome b and 12S rRNA mitochondrial sequences // Mol. Biol. Evol. 1997. V.14. P.550–559. Одновременный анализ молекулярных и морфологических данных, позволяющий учесть и вымершие виды, также не поддерживал гипотезу, согласно которой ветвь китов отходит от ветви парнокопытных [2]O’Leary M.A., Geisler J.H. The position of Cetacea within mammalia: phylogenetic analysis of morphological data from extinct and extant taxa // Syst. Biol. 1999. V.48. P.455–490.



Рис. 1. Современные киты и их ближайший родственник из ныне живущих [13]Gatesy J., Geisler J.H., Chang J. et al. A phylogenetic blueprint for a modern whale // Mol. Phylogenet. Evol. 2013. V.66. P.479–506.Doi:10.1016/j.ympev.2012.10.012 Изображены представители двух подотрядов китов: голубой кит (а; Balaenoptera musculus) из усатых и кашалот (б; Physeter macrocephalus) из зубатых — и карликовый бегемот (в; Choeropsis liberiensis).
Точку в споре поставил анализ не отдельных генов, а геномов в целом. Определение полного митохондриального генома гиппопотама (Hippopotamus amphibius) показало его близкое родство с китами [12]Ursing B.M., Arnason U. Analyses of mitochondrial genomes strongly support a hippopotamus-whale clade // Proc. Biol. Sci. 1998. V.265. P.2251–2255.Doi:10.1098/rspb.1998.0567 (рис. 1). Аналогичным оказался результат исследования повторяющихся элементов в ядерных геномах. У китов, гиппопотамов и жвачных парнокопытных, но не у свиней и верблюдов обнаружили два семейства специфических повторов [14]Shimamura M., Yasue H., Ohshima K. et al. Molecular evidence from retroposons that whales form a clade within even-toed ungulates // Nature. 1997. V.388. P.666–670.Doi:10.1038/41759 причем их эволюцию, в частности появление новых вариантов повторов, проследили детально [15]Shimamura M., Abe H., Nikaido M. et al. Genealogy of families of SINEs in cetaceans and artiodactyls: the presence of a huge superfamily of tRNA(Glu)-derived families of SINEs // Mol. Biol. Evol. 1999. V.16. P.1046–1060. Кроме того, так как геномы очень велики, а вставки повторяющихся элементов — события относительно редкие, они почти никогда не появляются в одном месте в разных геномах независимо, а стало быть, дают возможность детально проследить эволюцию на относительно малых временах. Когда набралось достаточное количество данных, гипотеза о родстве китов и гиппопотамов была окончательно подтверждена [16]Nikaido M., Rooney A.P., Okada N. Phylogenetic relationships among cetartiodactyls based on insertions of short and long interpersed elements: hippopotamuses are the closest extant relatives of whales // Proc. Natl. Acad. Sci. USA. 1999. V.96. P.10261–10266. Сомнения, касающиеся методики построения филогенетических деревьев по отдельным белкам, разрешились благодаря тому, что увеличение набора данных, например, при анализе 121 однокопийного гена ядерного генома 21 представителя китопарнокопытных, сделало построенные деревья статистически устойчивыми [17]Zhou X., Xu S., Yang Y. et al. Phylogenomic analyses and improved resolution of Cetartiodactyla // Mol. Phylogenet. Evol. 2011. V.61. P.255–264.Doi:10.1016/j.ympev.2011.02.009Такой же результат дал анализ митохондриальных геномов 183 видов китопарнокопытных (включая 128 новосеквенированных), среди которых были представители всех известных семейств [18]Hassanin A., Delsuc F., Ropiquet A. et al. Pattern and timing of diversification of Cetartiodactyla (Mammalia, Laurasiatheria), as revealed by a comprehensive analysis of mitochondrial genomes // C. R. Biol. 2012. V.335. P.32–50.Doi:10.1016/j.crvi.2011.11.002Расхождение ветвей, ведущих к современным китам, произошло в раннем олигоцене (34–28 млн лет назад) или, по другим работам, 37–33 млн лет назад, тогда как расхождение предков китов и гиппопотамов датируется 55–50 млн лет назад [19]Thewissen J.G., Madar S.I. Ankle morphology of the earliest Cetaceans and its implications for the phylogenetic relations among ungulates // Syst. Biol. 1999. V.48. P.21–30.

Подоспели и новые палеонтологические данные (рис. 2). Родство китов и парнокопытных подтверждалось находками костей голени ранних китов [13]Gatesy J., Geisler J.H., Chang J. et al. A phylogenetic blueprint for a modern whale // Mol. Phylogenet. Evol. 2013. V.66. P.479–506.Doi:10.1016/j.ympev.2012.10.012 Был описан род Indohyus — парнокопытных, образующих ближайшую сестринскую группу по отношению к китам после их разделения с гиппопотамами [20]Thewissen J.G., Cooper L.N., Clementz M.T. et al. Whales originated from aquatic artiodactyls in the Eocene epoch of India // Nature. 2007. V.450. P.1190–1194.Doi:10.1038/nature06343 Скелеты сухопутных предшественников китов, Ichthyolestes pinfoldi (размером с лису) и Pakicetus attocki (размером с волка), явно больше походили на скелеты парнокопытных, чем мезонихий, хотя морфологический анализ не подтверждал родства с гиппопотамами [21]Thewissen J.G., Williams E.M., Roe L.J. et al. Skeletons of terrestrial cetaceans and the relationship of whales to artiodactyls // Nature. 2001. V.413. P.277–281.Doi:10.1038/35095005 Однако при повторном анализе тех же скелетов ветвь гиппопотамов и китов была восстановлена [22]Geisler J.H., Theodor J.M. Hippopotamus and whale phylogeny // Nature. 2009. V.458. E1–E4.Doi:10.1038/nature07776 Строение конечностей еще двух предков китов, Artiocetus clavis и Rodhocetus balochistanensis, также имеет общие черты со строением конечностей парнокопытных, но не мезонихий [23]Gingerich P.D., Haq M., Zalmout I.S. et al. Origin of whales from early artiodactyls: hands and feet of Eocene Protocetidae from Pakistan // Science. 2001. V.293. P.2239–2242.Doi:10.1126/science.1063902 Построение филогенетических деревьев, учитывающих одновременно и геномные последовательности, и морфологические признаки современных китов, их предков и представителей вымерших линий, позволило датировать основные анатомические и физиологические изменения, привязав их к внутренним узлам филогенетического дерева [19]Thewissen J.G., Madar S.I. Ankle morphology of the earliest Cetaceans and its implications for the phylogenetic relations among ungulates // Syst. Biol. 1999. V.48. P.21–30.[24]McGowen M.R., Gatesy J., Wildman D.E. Molecular evolution tracks macroevolutionary transitions in Cetacea // Trends Ecol. Evol. 2014. V.29. P.336–346.Doi:10.1016/j.tree.2014.04.001 (рис. 3).
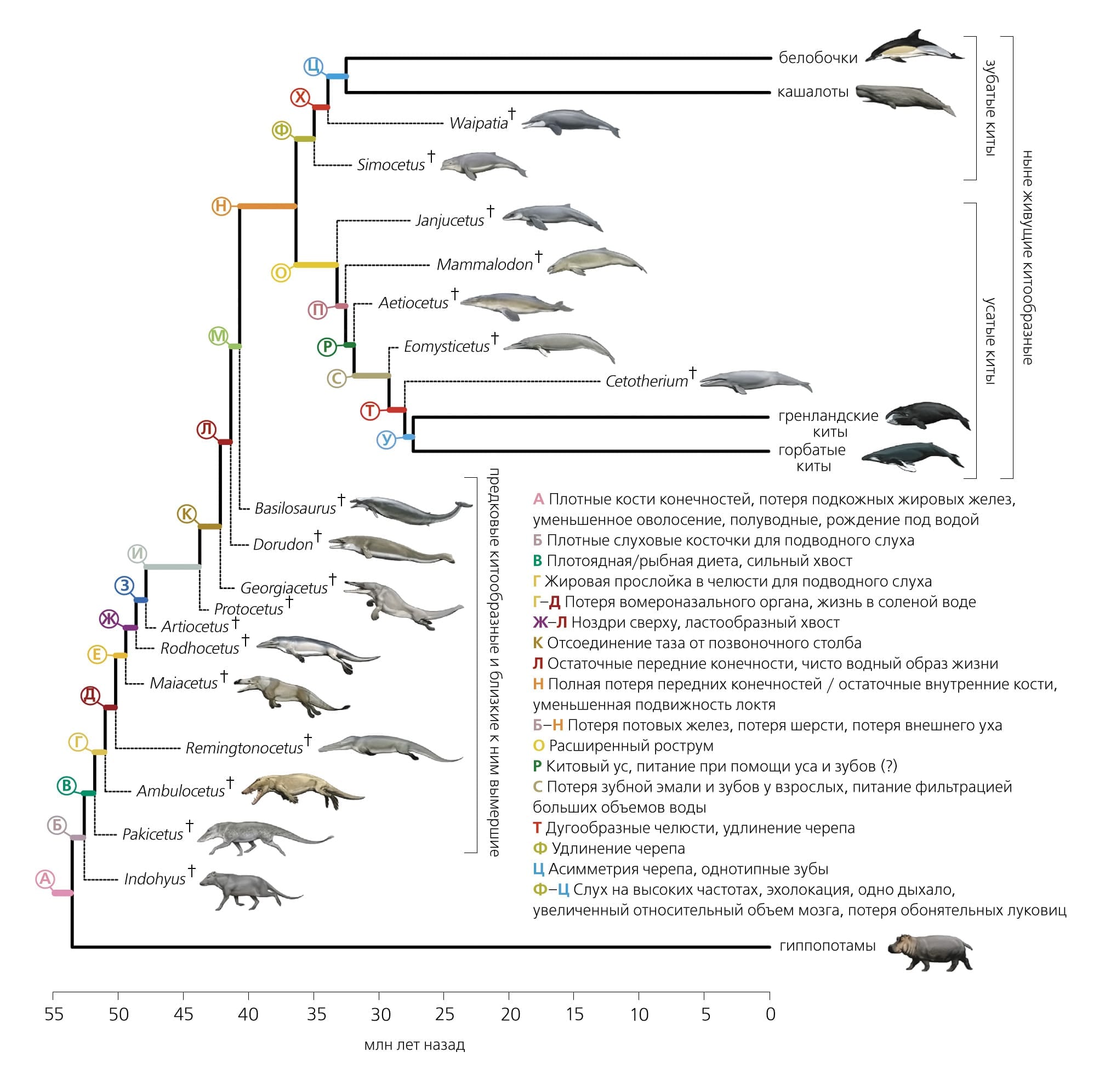
Затем был проведен сравнительный анализ отдельных генов различных видов китов и других млекопитающих. Кроме того, были проанализированы полные геномы представителей двух подотрядов ныне живущих китов: зубатого кита — китайского речного дельфина, или байцзи(*)До 1900 г. популяция составляла несколько тысяч особей, однако в 2007 г. вид уже был объявлен исчезнувшим. (Lipotes vexillifer) [25]Zhou X., Sun F., Xu S. et al. Baiji genomes reveal low genetic variability and new insights into secondary aquatic adaptations // Nat. Commun. 2013. V.4. P.2708.Doi:10.1038/ncomms3708 и усатого кита — малого полосатика (Balaenoptera acutorostrata) [26]Yim H.S., Cho Y.S., Guang X. et al. Minke whale genome and aquatic adaptation in cetaceans // Nat. Genet. 2014. V.46. P.88–92.Doi:10.1038/ng.2835 Было также проведено сравнение больших фрагментов геномов ряда других китообразных: дельфина-бутылконоса (Tursiops truncatus), финвала (Balaenoptera physalus), бесперой морской свиньи (Neophocaena phocaenoides) [26]Yim H.S., Cho Y.S., Guang X. et al. Minke whale genome and aquatic adaptation in cetaceans // Nat. Genet. 2014. V.46. P.88–92.Doi:10.1038/ng.2835 Все это в совокупности позволило описать паттерн эволюции — положительный отбор (поощряющий изменения; оценивается как отношение потока значимых замен в последовательности гена к синонимичным) — и, наоборот, потерю функций генов вследствие инактивирующих мутаций (сдвигов рамки считывания, нонсенс-мутаций) и сопоставить наблюдаемые события, происходившие на молекулярном уровне, с макроскопическими изменениями в анатомии и физиологии животных.
Миоглобин: киты учатся нырять
Кислород в мышцах запасается в комплексе с белком миоглобином, который гомологичен гемоглобину крови, но эволюционно разошелся с ним уже очень давно. Погружение под воду на длительное время требует больших запасов кислорода, а значит, и большей концентрации миоглобина. Действительно, концентрация миоглобина у китов в 10–20 раз выше, чем у их наземных родственников [27]Kooyman G.L., Ponganis P.J. The physiological basis of diving to depth: birds and mammals // Annu. Rev. Physiol. 1998. V.60. P.19–32. Более того, концентрация миоглобина в скелетных мышцах выше у тех китов, у которых больше интервал между вдохами, и выше у взрослых особей по сравнению с детенышами, что, по-видимому, отражает постепенное уменьшение частоты дыхания с возрастом [28]Dolar M.L., Suarez P., Ponganis P.J. et al. Myoglobin in pelagic small cetaceans // J. Exp. Biol. 1999. V.202. P.227–236. Аналогичный результат был получен и при анализе большего числа видов [29]Helbo S., Fago A. Functional properties of myoglobins from five whale species with different diving capacities // J. Exp. Biol. 2012. V.215. P.3403–3410.Doi:10.1242/jeb.073726 Учет концентрации миоглобина и массы тела позволяет объяснить большую часть вариаций в продолжительности погружений [30]Noren S.R., Williams T.M. Body size and skeletal muscle myoglobin of cetaceans: adaptations for maximizing dive duration // Comp. Biochem. Physiol. A Mol. Integr. Physiol. 2000. V.126. P.181–191.Doi:10.1016/S1095-6433(00)00182-3 В то же время не было обнаружено никакой связи между способностью миоглобина связывать кислород и длительностью и глубиной погружений [29]Helbo S., Fago A. Functional properties of myoglobins from five whale species with different diving capacities // J. Exp. Biol. 2012. V.215. P.3403–3410.Doi:10.1242/jeb.073726
При чрезмерной концентрации миоглобина его молекулы агрегируют, теряют стабильность и становятся нефункциональными. Положительный отбор, действующий на миоглобин китов, привел к увеличению его стабильности за счет гидрофобных взаимодействий и образования дополнительных водородных связей. При этом наблюдается корреляция между стабильностью и концентрацией миоглобина у разных видов [31]Dasmeh P., Kepp K.P. Bridging the gap between chemistry, physiology, and evolution: quantifying the functionality of sperm whale myoglobin mutants // Comp. Biochem. Physiol. A Mol. Integr. Physiol. 2012. V.161. P.9–17.Doi:10.1016/j.cbpa.2011.07.027 Приспособление к глубоководному нырянию произошло у общего предка зубатых и усатых китов до расхождения их ветвей. Способность погружаться на большие глубины усилилась у кашалота и несколько ослабла у дельфинов и морских свиней [32]Holm J., Dasmeh P., Kepp K.P. Tracking evolution of myoglobin stability in cetaceans using experimentally calibrated computational methods that account for generic protein relaxation // Biochim. Biophys. Acta. 2016. V.1864. P.825–834.Doi:10.1016/j.bbapap.2016.04.004 При этом у разных семейств наблюдаются параллельные замены [33]Nery M.F., Arroyo J.I., Opazo J.C. Accelerated evolutionary rate of the myoglobin gene in long-diving whales // J. Mol. Evol. 2013. V.76. P.380–387.Doi:10.1007/s00239-013-9572-1 Следует отметить, однако, что списки конкретных позиций, находящихся под действием положительного отбора, в разных статьях могут довольно значительно различаться вследствие неодинаковости выборок и вычислительных моделей. Эти различия, кстати, представляют собой интересную тему для будущего исследования.
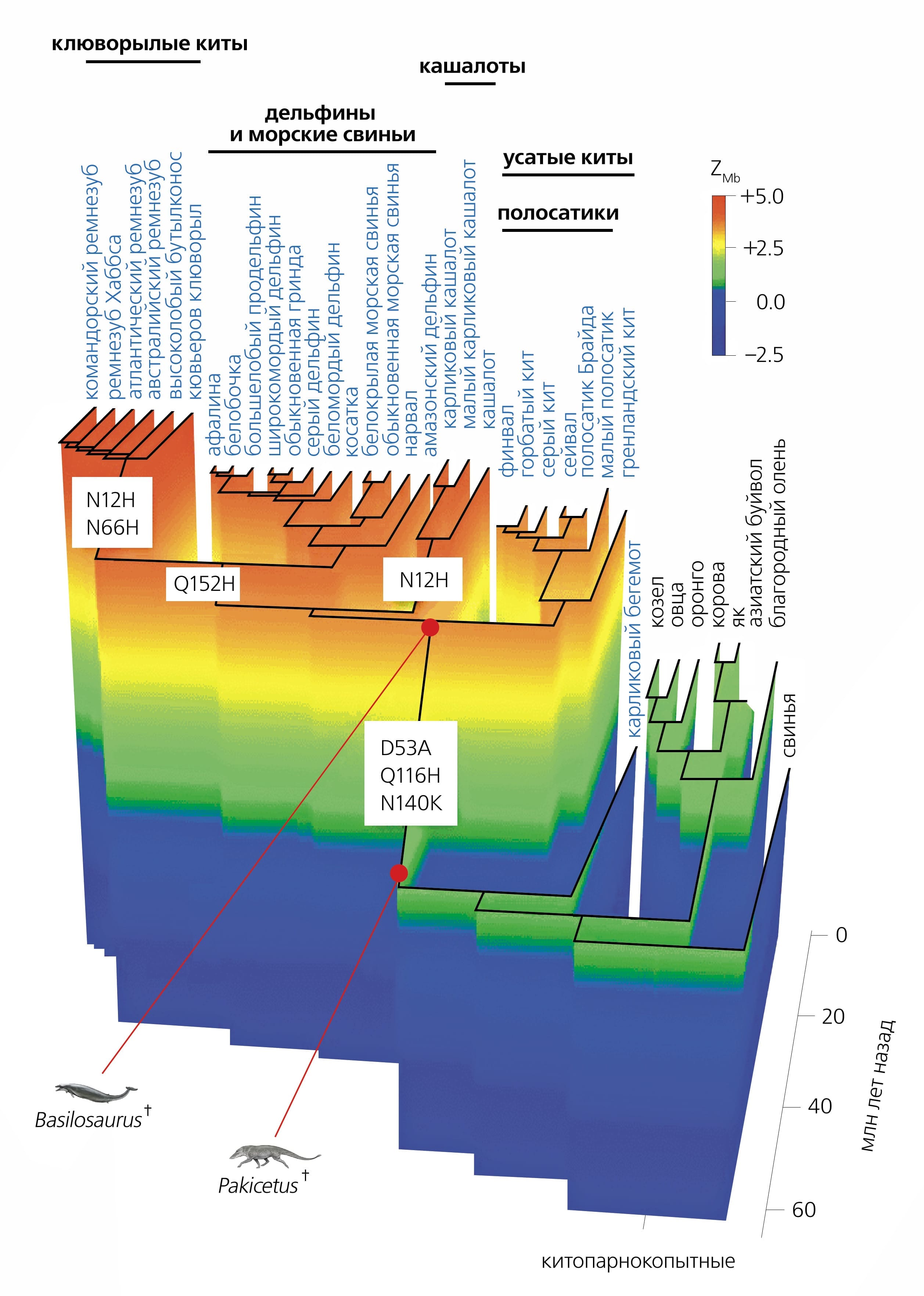
Совокупность этих наблюдений позволяет предсказывать продолжительность погружений для предков китов [34]Mirceta S., Signore A.V., Burns J.M. et al. Evolution of mammalian diving capacity traced by myoglobin net surface charge // Science. 2013. V.340. P.1234192.Doi:10.1126/science.1234192 В самом деле, наблюдается линейная зависимость между поверхностным зарядом и максимальной концентрацией миоглобина в скелетных мышцах, а последняя линейно связана с логарифмом массы тела и логарифмом продолжительности вдоха. Массу тела вымершего животного можно оценить по его скелету. Для того чтобы узнать поверхностный заряд миоглобина у предкового вида, надо реконструировать аминокислотную последовательность белка. Пространственная структура миоглобина очень консервативна, а потому, зная последовательность предкового белка, легко смоделировать его структуру и подсчитать поверхностный заряд стандартными методами (рис. 4). Итак, теперь мы знаем все, что нужно, чтобы увидеть, как у предков китов постепенно увеличивалась максимальная продолжительность непрерывного пребывания под водой (рис. 5).
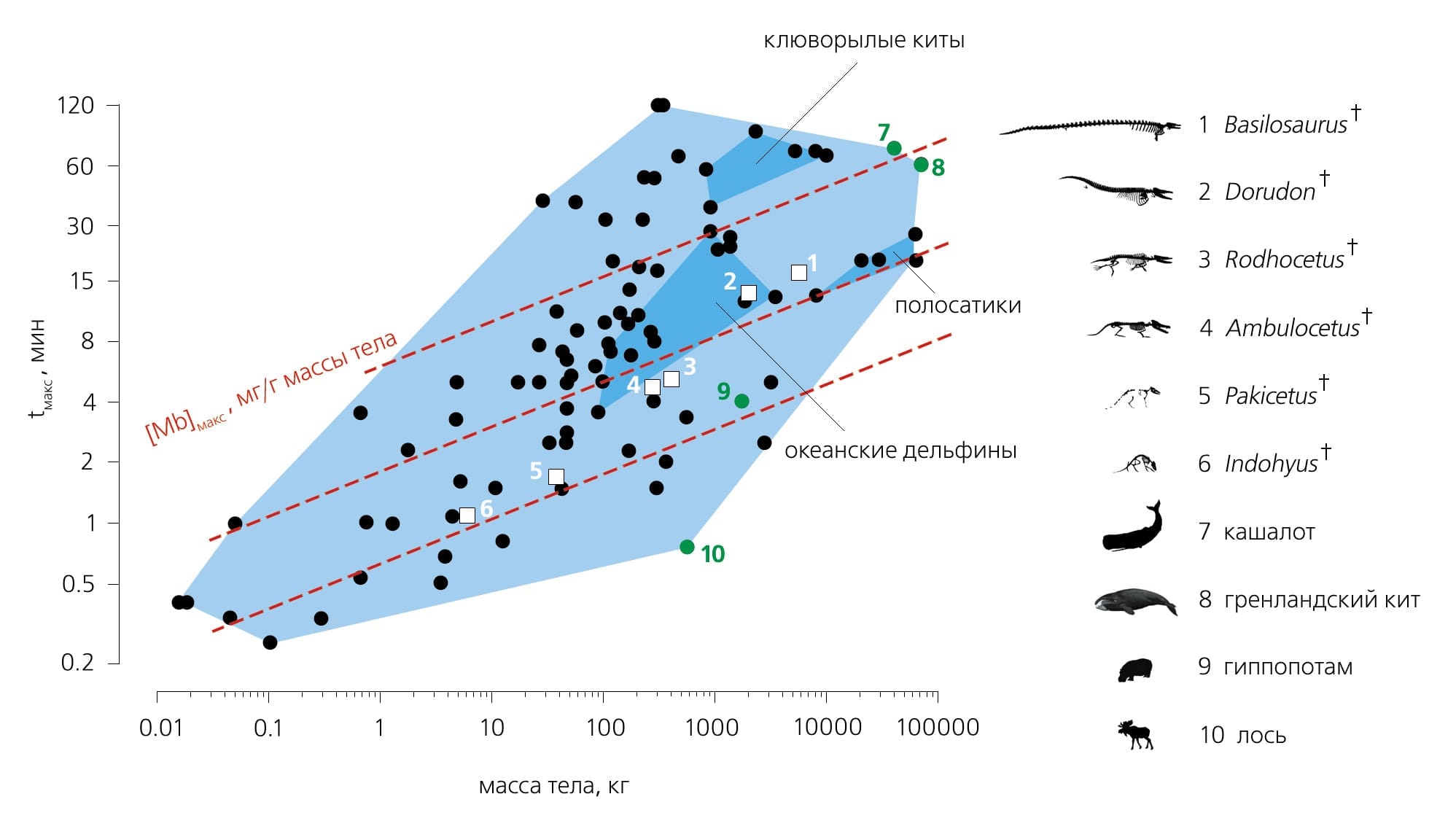
Дополнительные примеры молекулярных адаптаций к недостатку кислорода будут приведены далее, при обсуждении конкретных физиологических систем.
Мозг и органы чувств
Переход к жизни в водной среде сильно поменял органы чувств китов: зрение приспособилось к другому освещению; сильно редуцировалось (у усатых китов) или совсем исчезло (у зубатых) обоняние; появилась эхолокация в ультразвуковом диапазоне. При этом киты обладают самым большим среди млекопитающих головным мозгом, и у них, по всей видимости, имеется сложная система коммуникации. Многие исследователи пытались найти генетические корреляты этих изменений. Ряд наблюдений представляется весьма многообещающим, хотя, безусловно, уровня наших знаний о сложных регуляторных взаимодействиях в эмбриональном и эволюционном развитии млекопитающих недостаточно для того, чтобы говорить о прямых причинно-следственных связях.
Пожалуй, одна из основных проблем, связанных с работой мозга у китов, — необходимость поддерживать высокий уровень метаболизма, а значит, и потребления кислорода при значительном интервале между вдохами. Одним из решений оказалось повышение (в 4–15 раз) уровня экспрессии гена нейроглобина — белка, который участвует в запасании кислорода и защищает от его агрессивных форм ткани мозга [35]Schneuer M., Flachsbarth S., Czech-Damal N.U. et al. Neuroglobin of seals and whales: evidence for a divergent role in the diving brain // Neuroscience. 2012. V.223. P.35–44.Doi:10.1016/j.neuroscience.2012.07.052 Примечательно, что у ластоногих наблюдается другое приспособление — синтез нейроглобина не в нейронах, а в астроцитах.
Следы действия положительного отбора были найдены в гене микроцефалина, который контролирует размер мозга у млекопитающих, однако сила отбора не коррелировала с размером мозга современных китов [36]McGowen M.R., Montgomery S.H., Clark C. et al. Phylogeny and adaptive evolution of the brain-development gene microcephalin (MCPH1) in cetaceans // BMC Evol. Biol. 2011. V.11. P.98.Doi:10.1186/1471-2148-11-98 В линии, ведущей к дельфинам, 27 из 228 генов, находящихся под действием положительного отбора, оказались связанными с нервной системой (у человека мутации в этих генах вызывают проблемы с интеллектом и влияют на синаптическую пластичность и сон) [37]McGowen M.R., Grossman L.I., Wildman D.E. Dolphin genome provides evidence for adaptive evolution of nervous system genes and a molecular rate slowdown // Proc. Biol. Sci. 2012. V.279. P.3643–3651.Doi:10.1098/rspb.2012.0869
Последствия положительного отбора обнаружились также в генах, кодирующих белки, важные для развития слуховой системы: престин (моторный белок, который вырабатывается в волосковых наружных клетках внутреннего уха) [38]Li Y., Liu Z., Shi P. et al. The hearing gene Prestin unites echolocating bats and whales // Curr. Biol. 2010. V.20. R55–R56.Doi:10.1016/j.cub.2009.11.042[39]Liu Y., Cotton J.A., Shen B. et al. Convergent sequence evolution between echolocating bats and dolphins // Curr. Biol. 2010. V.20. R53–R54.Doi:10.1016/j.cub.2009.11.058 трансмембранный белок TMC1 (механоэлектрический передатчик сигнала волосковых клеток внутреннего уха) и пежвакин (белок, функция которого не вполне ясна; мутации в соответствующем гене вызывают врожденную потерю слуха) [40]Davies K.T., Cotton J.A., Kirwan J.D. et al. Parallel signatures of sequence evolution among hearing genes in echolocating mammals: an emerging model of genetic convergence // Heredity. 2012. V.108. P.480–489.Doi:10.1038/hdy.2011.119 клаудин-14 (белок щелевых контактов, работающий в кортиевом органе внутреннего уха) [41]Xu H., Liu Y., He G. et al. Adaptive evolution of tight junction protein claudin-14 in echolocating whales // Gene. 2013. V.530. P.208–214.Doi:10.1016/j.gene.2013.08.034 Особенно интересно то, что во многих случаях параллельные изменения происходили и в генах летучих мышей, которые, как известно, используют эхолокацию. Иногда, как в случае генов, кодирующих кадерин-23 и его лиганд протокадерин-15 (белки, участвующие в образовании волосковых пучков), а также отоферин (белок, отвечающий на сигнал во внутренних волосковых клетках), ускорения эволюции не наблюдается, но параллелизм изменений у зубатых китов и летучих мышей свидетельствует о действии положительного отбора [42]Shen Y.Y., Liang L., Li G.S. et al. Parallel evolution of auditory genes for echolocation in bats and toothed whales // PLoS Genet. 2012. V.8. e1002788.Doi:10.1371/journal.pgen.1002788 Логично предположить, что изменившиеся белки, кодируемые этими генами, как раз и позволяют слышать в ультразвуковом диапазоне. И действительно, прямая связь между изменениями аминокислотной последовательности белка и способностью воспринимать очень высокий ультразвук была показана для престина [43]Liu Y., Rossiter S.J., Han X. et al. Cetaceans on a molecular fast track to ultrasonic hearing // Curr. Biol. 2010. V.20. P.1834–1839.Doi:10.1016/j.cub.2010.09.008 Среди 368 генов, находящихся под действием положительного отбора у дельфинов, существенную долю составляют те, которые связаны со слухом [44]Sun Y.B., Zhou W.P., Liu H.Q. et al. Genome-wide scans for candidate genes involved in the aquatic adaptation of dolphins // Genome Biol. Evol. 2013. V.5. P.130–139.Doi:10.1093/gbe/evs123
Как и следовало ожидать, утрата обоняния на молекулярном уровне проявляется как потеря генов, кодирующих рецепторы запахов. В результате мутаций они становятся псевдогенами и перестают кодировать функциональные белки, хотя и остаются в геноме [45]McGowen M.R., Clark C., Gatesy J. The vestigial olfactory receptor subgenome of odontocete whales: phylogenetic congruence between gene-tree reconciliation and supermatrix methods // Syst. Biol. 2008. V.57. P.574–590.Doi:10.1080/10635150802304787 То же касается и вкуса: анализ 10 генов, кодирующих полный репертуар рецепторов T2R для горького, и трех генов, кодирующих все типы рецепторов T1R для сладкого и умами, показал у 12 китов массивную потерю генов. Кроме того, ген, кодирующий рецептор PKD2L1 для кислого, — по-видимому, псевдоген, в то время как рецепторы для соленого функциональны [46]Feng P., Zheng J., Rossiter S.J. et al. Massive losses of taste receptor genes in toothed and baleen whales // Genome Biol. Evol. 2014. V.6. P.1254–1265.Doi:10.1093/gbe/evu095 В другой работе была показана потеря всех рецепторов, за исключением того, который воспринимает соленое [47]Zhu K., Zhou X., Xu S. The loss of taste genes in cetaceans // BMC Evol. Biol. 2014. V.14. P.218.Doi:10.1186/s12862-014-0218-8 Обеднение или утрата вкуса и обоняния случились у общего предка современных китов, что согласуется и с данными сравнительной анатомии [48]Kishida T., Thewissen J., Hayakawa T. et al. Aquatic adaptation and the evolution of smell and taste in whales // Zoological Lett. 2015. V.13. P.9.Doi:10.1186/s40851-014-0002-z
Так как свет с разной длиной волны проникает в толщу воды с разной эффективностью, одним из приспособлений к водному образу жизни служит смещение максимума спектра поглощения зрительных пигментов. Так, у дельфина-бутылконоса этот максимум сдвинут в сторону коротких длин волн, к синей части спектра, у палочкового пигмента (родопсина) и колбочкового (длинноволнового опсина), причем исследования мутантных пигментов позволили установить конкретные аминокислотные остатки, отвечающие за такой сдвиг [49]Fasick J.I., Robinson P.R. Mechanism of spectral tuning in the dolphin visual pigments // Biochemistry. 1998. V.37. P.433–438.Doi:10.1021/bi972500j В то же время у водных млекопитающих, живущих на мелководье, такой адаптации не наблюдается, а спектры поглощения зрительных пигментов совпадают с аналогичными спектрами наземных видов [50]Fasick J.I., Robinson P.R. Spectral-tuning mechanisms of marine mammal rhodopsins and correlations with foraging depth // Vis. Neurosci. 2000. V.17. P.781–788. В гене родопсина у китов положительный отбор затрагивает именно те аминокислотные остатки, мутации в которых приводят к сдвигу максимума поглощения в синюю часть спектра, ведь на глубину, куда ныряют эти животные, лучше всего проникает именно сине-зеленый свет [51]Dungan S.Z., Kosyakov A., Chang B.S. Spectral tuning of killer whale (Orcinus orca) rhodopsin: evidence for positive selection and functional adaptation in a cetacean visual pigment // Mol. Biol. Evol. 2016. V.33. P.323–336.Doi:10.1093/molbev/msv217 С одной стороны, у китов наблюдается также сдвиг максимума поглощения у длинноволнового опсина в сторону синего света [52]Newman L.A., Robinson P.R. Cone visual pigments of aquatic mammals // Vis. Neurosci. 2005. V.22. P.873–879.Doi:10.1017/S0952523805226159 С другой — в их геномах есть и псевдоген коротковолнового опсина, нефункциональный из-за многочисленных мутаций, приводящих к сдвигу рамки считывания. Согласно результатам разных исследований, потеря функции случилась или до разделения на зубатых и усатых китов [53]Fasick J.I., Cronin T.W., Hunt D.M. et al. The visual pigments of the bottlenose dolphin (Tursiops truncatus) // Vis. Neurosci. 1998. V.15. P.643–651.[54]Levenson D.H., Dizon A. Genetic evidence for the ancestral loss of short-wavelength-sensitive cone pigments in mysticete and odontocete cetaceans // Proc. Biol. Sci. 2003. V.270. P.673–679.Doi:10.1098/rspb.2002.2278 или независимо у тех и других [55]Meredith R.W., Gatesy J., Emerling C.A. et al. Rod monochromacy and the coevolution of cetacean retinal opsins // PLoS Genet. 2013. V.9. e1003432.Doi:10.1371/journal.pgen.1003432 Комбинация утраты коротковолнового опсина и сдвига максимума поглощения в коротковолновую часть спектра у длинноволнового опсина с родопсином выглядит парадоксально — возможно, дело в относительной датировке этих событий. Отмечают также, что переход к палочковому монохроматизму не сказался на гистологическом строении сетчатки и связанных с ней сигнальных структур, в частности биполярных клеток, взаимодействующих с колбочками, и синапсов колбочек [56]Schweikert L.E., Fasick J.I., Grace M.S. Evolutionary loss of cone photoreception in balaenid whales reveals circuit stability in the mammalian retina // J. Comp. Neurol. 2016. V.524. P.2873–2885.Doi:10.1002/cne.23996 но, опять же, странно говорить о палочковом монохроматизме при сохранении даже одного типа опсина в колбочках. Следовало бы проверить их функциональность, ведь опыты на цветовосприятие у дельфинов кажутся вполне осуществимыми.
Другие функциональные системы
В любой быстро развивающейся области результаты конкретных исследований в деталях могут друг другу противоречить. Скажем, количество генов, находящихся под действием положительного отбора в геноме дельфина, разные исследователи оценили как 228 [37]McGowen M.R., Grossman L.I., Wildman D.E. Dolphin genome provides evidence for adaptive evolution of nervous system genes and a molecular rate slowdown // Proc. Biol. Sci. 2012. V.279. P.3643–3651.Doi:10.1098/rspb.2012.0869 368 [44]Sun Y.B., Zhou W.P., Liu H.Q. et al. Genome-wide scans for candidate genes involved in the aquatic adaptation of dolphins // Genome Biol. Evol. 2013. V.5. P.130–139.Doi:10.1093/gbe/evs123 и 376 [57]Nery M.F., González D.J., Opazo J.C. How to make a dolphin: molecular signature of positive selection in cetacean genome // PLoS One. 2013. V.8. e65491.Doi:10.1371/journal.pone.0065491 Функциональные системы, в которых особенно часто работают продукты таких генов, включают нервную и сердечно-сосудистую системы, энергетический метаболизм (в особенности глюкозы и липидов), развитие кожи и легких [34]Mirceta S., Signore A.V., Burns J.M. et al. Evolution of mammalian diving capacity traced by myoglobin net surface charge // Science. 2013. V.340. P.1234192.Doi:10.1126/science.1234192 транспорт и локализацию липидов, АТФазную активность, слух и мышечные сокращения [44]Sun Y.B., Zhou W.P., Liu H.Q. et al. Genome-wide scans for candidate genes involved in the aquatic adaptation of dolphins // Genome Biol. Evol. 2013. V.5. P.130–139.Doi:10.1093/gbe/evs123 сегментацию и развитие мезодермы [57]Nery M.F., González D.J., Opazo J.C. How to make a dolphin: molecular signature of positive selection in cetacean genome // PLoS One. 2013. V.8. e65491.Doi:10.1371/journal.pone.0065491 В то же время даже такие наборы генов, полученные разными методами (поучительно было бы сравнить полные наборы генов из этих работ), имеют много общего и вполне могут быть интерпретированы в терминах физиологических и анатомических адаптаций. Многие различия могут быть связаны не с изменением генов, а точнее, кодируемых ими белков, а с изменением работы генов — включением, выключением и интенсивностью экспрессии на разных стадиях развития. Однако возможности современных биоинформатики и геномики для оценки уровня экспрессии генов по анализу регуляторных мотивов в последовательностях ДНК не очень велики (для эукариот, с бактериями все проще), а ожидать массового потока данных по транскриптомам эмбрионов китов различных видов (для сравнительного анализа) было бы слишком оптимистично.
Анатомия
Одной из самых ярких анатомических адаптаций китов стало развитие ласт, сопровождавшееся существенной редукцией скелета конечностей. Парадоксальным образом этому процессу предшествовало восстановление пальцев, утраченных копытными предшественниками. И действительно, ген Hox12, контролирующий у млекопитающих развитие конечностей и пальцев, находится у китов под действием сильного положительного отбора, а удлинение полиаланинового тракта в гене Hox13 из той же регуляторной сети, наблюдающееся у китов, напоминает аналогичную мутацию у человека, ведущую к полидактилии [58]Wang Z., Yuan L., Rossiter S.J. et al. Adaptive evolution of 5’HoxD genes in the origin and diversification of the cetacean flipper // Mol. Biol. Evol. 2009. V.26. P.613–622.Doi:10.1093/molbev/msn282 У дельфинов обнаружились следы действия положительного отбора в гене Hox9, от которого также зависит развитие передних конечностей [59]Liang L., Shen Y.Y., Pan X.W. et al. Adaptive evolution of the Hox gene family for development in bats and dolphins // PLoS One. 2013. V.8. e65944.Doi:10.1371/journal.pone.0065944 При тотальном анализе генома дельфина последствия положительного отбора были найдены в генах, отвечающих за сегментацию тела [57]Nery M.F., González D.J., Opazo J.C. How to make a dolphin: molecular signature of positive selection in cetacean genome // PLoS One. 2013. V.8. e65491.Doi:10.1371/journal.pone.0065491 мышечные сокращения [44]Sun Y.B., Zhou W.P., Liu H.Q. et al. Genome-wide scans for candidate genes involved in the aquatic adaptation of dolphins // Genome Biol. Evol. 2013. V.5. P.130–139.Doi:10.1093/gbe/evs123 и развитие сердечно-сосудистой системы, легких и кожи [37]McGowen M.R., Grossman L.I., Wildman D.E. Dolphin genome provides evidence for adaptive evolution of nervous system genes and a molecular rate slowdown // Proc. Biol. Sci. 2012. V.279. P.3643–3651.Doi:10.1098/rspb.2012.0869
Потеря оволосения может быть связана с изменением работы двух ключевых регуляторов роста волосяных фолликулов — генов Hr и FGF5 [60]Chen Z., Wang Z., Xu S. et al. Characterization of hairless (Hr) and FGF5 genes provides insights into the molecular basis of hair loss in cetaceans // BMC Evol. Biol. 2013. V.13. P.34.Doi:10.1186/1471-2148-13-34 Удивительно, но изменения произошли в противоположную сторону: FGF5 быстро эволюционировал, а Hr превратился в псевдоген. Впрочем, это согласуется с сигнальной ролью этих факторов: изменившийся FGF5, видимо, останавливает рост волос и переводит фолликулы в стадию восстановления, а отсутствие работающего Hr приводит к нарушениям в этой стадии. Псевдогенами стали также многие гены β-кератинов, основных белков волос [61]Nery M.F., Arroyo J.I., Opazo J.C. Increased rate of hair keratin gene loss in the cetacean lineage // BMC Genomics. 2014. V.15. P.869.Doi:10.1186/1471-2164-15-869 У усатых китов потеря зубов, похоже, сопровождалась превращением генов, кодирующих структурные белки зубной эмали энамелин и амелобластин, в псевдогены [62]Deméré T.A., McGowen M.R., Berta A. et al. Morphological and molecular evidence for a stepwise evolutionary transition from teeth to baleen in mysticete whales // Syst. Biol. 2008. V.57. P.15–37.Doi:10.1080/10635150701884632
Физиология
В связи с переходом китов к морскому образу жизни естественно было ожидать приспособления к новой среде у аквапоринов и других белков, участвующих в осмотической регуляции. Анализ семи аквапориновых генов показал действие положительного отбора на ген аквапорина-2, специфического для ветви китов. У других аквапоринов действие положительного отбора завершилось до отделения китов от парнокопытных [63]São Pedro S.L., Alves J.M., Barreto A.S. et al. Evidence of positive selection of aquaporins genes from Pontoporia blainvillei during the evolutionary process of cetaceans // PLoS One. 2015. V.10. e0134516.Doi:10.1371/journal.pone.0134516 Изменения в аквапорине-2 и транспортере мочевины UT-A2 могут быть связаны с транспортом воды и мочевины, способствуя более высокой концентрации мочевины в моче и экономии воды [64]Wang J., Yu X., Hu B. et al. Physicochemical evolution and molecular adaptation of the cetacean osmoregulation-related gene UT-A2 and implications for functional studies // Sci. Rep. 2015. V.5. P.8795.Doi:10.1038/srep08795 В свою очередь, изменения в ангиотензиногене и ангиотензин-конвертирующем ферменте, по-видимому, связаны с необходимостью поддержания водно-солевого баланса в гиперосмотической среде [65]Xu S., Yang Y., Zhou X. et al. Adaptive evolution of the osmoregulation-related genes in cetaceans during secondary aquatic adaptation // BMC Evol. Biol. 2013. V.13. P.189.Doi:10.1186/1471-2148-13-189
Молекулярным коррелятом приспособления к более эффективному переносу кислорода стали параллельные замены и положительный отбор, который действует не только на миоглобин, но и на α- и β-гемоглобины (перенос кислорода в крови), а также на регуляторы тонуса сосудов и сокращения их стенок: эндотелины-1, -2 и -3, рецепторы эндотелина типов A и B, адренергический рецептор α-1D и антидиуретический гормон [66]Tian R., Wang Z., Niu X. et al. Evolutionary genetics of hypoxia tolerance in cetaceans during diving // Genome Biol. Evol. 2016. V.8. P.827–839.Doi:10.1093/gbe/evw037
Положительный отбор, действующий на мембранную часть сурфактантного белка C, мог способствовать приспособлению легких к циклу сжатий–раздуваний, важному для глубокого погружения при нырянии [67]Foot N.J., Orgeig S., Donnellan S. Positive selection in the N-terminal extramembrane domain of lung surfactant protein C (SP-C) in marine mammals // J. Mol .Evol. 2007. V.65. P.12–22.Doi:10.1007/s00239-006-0083-1
У дельфина последствия положительного отбора обнаружились в генах, участвующих в транспорте и метаболизме глюкозы и липидов, в том числе в митохондриях [37]McGowen M.R., Grossman L.I., Wildman D.E. Dolphin genome provides evidence for adaptive evolution of nervous system genes and a molecular rate slowdown // Proc. Biol. Sci. 2012. V.279. P.3643–3651.Doi:10.1098/rspb.2012.0869[44]Sun Y.B., Zhou W.P., Liu H.Q. et al. Genome-wide scans for candidate genes involved in the aquatic adaptation of dolphins // Genome Biol. Evol. 2013. V.5. P.130–139.Doi:10.1093/gbe/evs123 а также в трансмембранной α-спирали второй субъединицы митохондриальной NADH-дегидрогеназы, возможно, задействованной в поддержании осмотического баланса митохондрий; в последнем случае наблюдались также многочисленные параллельные замены предкового аланина или валина на тирозин [68]Caballero S., Duchêne S., Garavito M.F. et al. Initial evidence for adaptive selection on the NADH subunit two of freshwater dolphins by analyses of mitochondrial genomes // PLoS One. 2015. V.10. e0123543.Doi:10.1371/journal.pone.0123543 Замены в гене цитохрома b могли повлиять на эффективность энергетических процессов [69]McClellan D.A., Palfreyman E.J., Smith M.J. et al. Physicochemical evolution and molecular adaptation of the cetacean and artiodactyl cytochrome b proteins // Mol. Biol. Evol. 2005. V.22. P.437–455.Doi:10.1093/molbev/msi028
Микробиом и иммунитет
Новая водная среда и рацион — рыба, планктон — требовали и совершенно иного набора симбиотических бактерий. Анализ бактериального сообщества дельфинов показал существенное число уникальных видов по сравнению с наземными родственниками [70]Bik E.M., Costello E.K., Switzer A.D. et al. Marine mammals harbor unique microbiotas shaped by and yet distinct from the sea // Nat. Commun. 2016. V.7. P.10516.Doi:10.1038/ncomms10516 У усатых китов обнаружилась смесь видов бактерий, характерных для хищников и травоядных [71]Sanders J.G., Beichman A.C., Roman J. et al. Baleen whales host a unique gut microbiome with similarities to both carnivores and herbivores // Nat. Commun. 2015. V.6. P.8285.Doi:10.1038/ncomms9285 Авторы объясняют это особенностями диеты: пути катаболизма белков и затем аминокислот, характерные для хищников, должны работать при питании рыбой, а пути катаболизма полисахаридов — при питании планктонными ракообразными, имеющими хитиновую оболочку. Впрочем, последнее предположение выглядит странно: катаболизм хитина, особенно на первых стадиях, существенно отличается от катаболизма полисахаридов, составляющих клеточную стенку растений, например целлюлозы, общая же часть этих метаболических путей относится к центральному метаболизму и присутствует практически у всех бактерий.
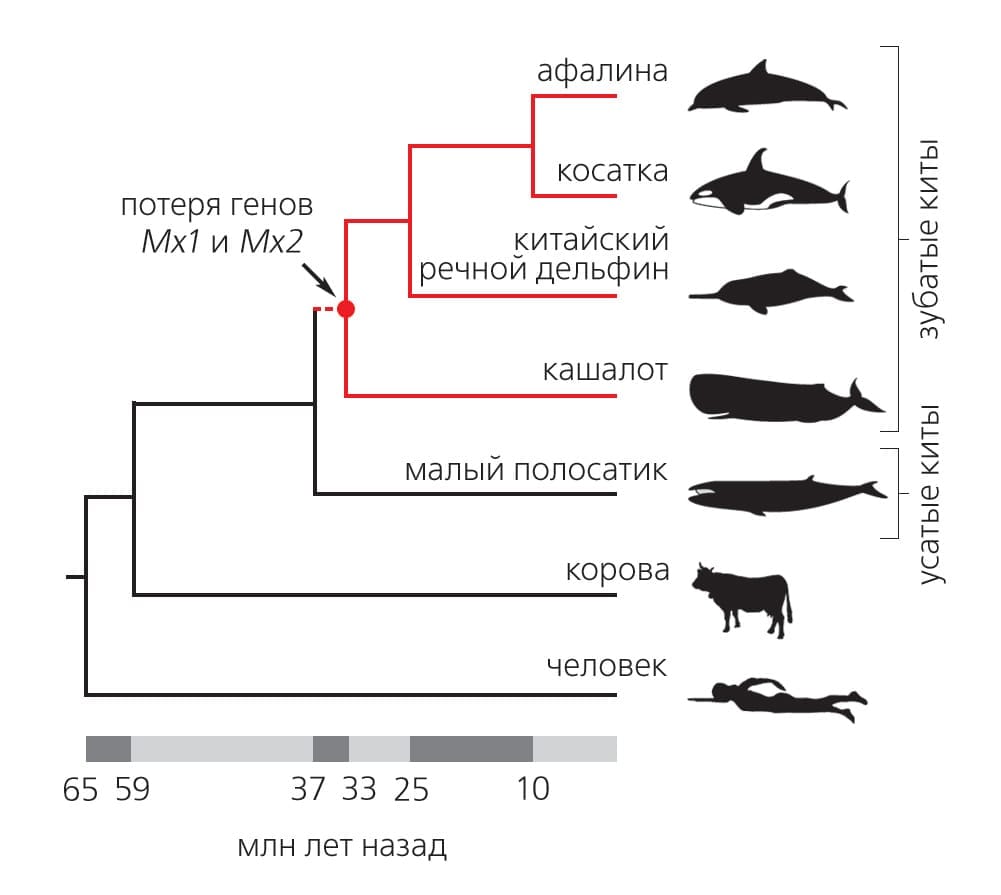
Как бы то ни было, быстрая адаптация к новым, морским, микробиомам потребовала и эволюции генов иммунной системы. Toll-подобные рецепторы, узнающие липополисахариды, формируют первую линию взаимодействия с бактериями. Оказалось, что некоторые участки гена TLR4, кодирующего одного из представителей семейства этих рецепторов, изменяются существенно быстрее, чем при случайном дрейфе, и в них происходят параллельные, т.е. одинаковые, замены в разных эволюционных линиях [72]Shen T., Xu S., Wang X. et al. Adaptive evolution and functional constraint at TLR4 during the secondary aquatic adaptation and diversification of cetaceans // BMC Evol. Biol. 2012. V.12. P.39.Doi:10.1186/1471-2148-12-39 Это служит свидетельством положительного отбора. Более того, выяснилось, что особенно сильно эволюция ускорилась во время перехода от сухопутного образа жизни к полуводному у общего предка китов и гиппопотамов и во время перехода к полностью водному образу жизни у предка китов. Моделирование пространственной структуры рецептора показывает, что приобретенные замены могут влиять на его взаимодействие с другими белками, участвующими в распознавании липополисахаридов, а стало быть, на иммунитет к грамотрицательным бактериям [73]Shishido R., Ohishi K., Suzuki R. et al. Cetacean Toll-like receptor 4 and myeloid differentiation factor 2, and possible cetacean-specific responses against gram-negative bacteria // Comp. Immunol. Microbiol. Infect. Dis. 2010. V.33. e89–e98.Doi:10.1016/j.cimid.2010.03.003 При этом у зубатых китов были потеряны гены Mx1 и Mx2, отвечающие за противовирусный иммунитет [74]Braun B.A., Marcovitz A., Camp J.G. et al. Mx1 and Mx2 key antiviral proteins are surprisingly lost in toothed whales // Proc. Natl. Acad. Sci. USA. 2015. V.30. P.8036–8040.Doi:10.1073/pnas.1501844112 (рис. 6).
* * *
Перечисленные в этом обзоре наблюдения носят достаточно отрывочный характер по трем причинам. Во-первых, нынешний уровень понимания того, как генетическая информация преобразуется в анатомические и физиологические особенности, явно недостаточен для систематического исследования функциональных систем организма и их эволюции. Во-вторых, для такого исследования не хватает данных — полностью секвенированных геномов ныне живущих китов, а тем более транскриптомов отдельных тканей и органов, желательно на разных стадиях развития. В-третьих, вполне вероятно, что разнообразия ныне живущих видов может оказаться недостаточно для полной эволюционной реконструкции событий, произошедших между разделением ветвей китов и гиппопотамов и разделением китов на основные современные ветви, ведь именно в эти 30 млн лет происходили основные морфологические изменения.
В заключение стоит отметить, что анализ геномов не только проливает свет на эволюцию китов, но и позволяет находить новые ныне живущие виды. В конце 1970-х годов японские китобои «поймали» восемь китов ранее неизвестного вида, похожих на финвалов. (Так деликатно написано в статье японских же авторов [75]Wada S., Oishi M., Yamada T.K. A newly discovered species of living baleen whale // Nature. 2003. V.426. P.278–281.Doi:10.1038/nature02103 на самом деле китов убили, потому что Япония — одна из немногих стран, до сих пор активно занимающаяся китобойным промыслом под вывеской научных исследований.) Туша животного того же вида была выброшена на берег в 1998 г. Спустя пять лет, в 2003 г., анализ геномных последовательностей и характерных анатомических особенностей позволил описать новый вид — полосатика Омуры (Balaenoptera omurai). Именно молекулярные свидетельства стали решающими в определении того, не относятся ли эти особи к уже известному виду — полосатикам Брайда (B. edeni). Одновременно разрешилось и существовавшее в литературе противоречие — относятся ли киты Брайда к одному виду или к двум (B. edeni и B. brydei). Оказалось, что к двум.
А самая свежая находка была сделана в июле этого года [76]Morin P.A., Baker C.S., Brewer R.S. et al. Genetic structure of the beaked whale genus Berardius in the North Pacific, with genetic evidence for a new species // Marine Mammal. Science. 2016.Doi:10.1111/mms.12345 До сих пор были известны два вида клюворылых китов рода Berardius — клюворыл Бэрда, или северный плавун, живущий в северной части Тихого океана, и клюворыл Арну, или южный плавун, из Антарктики. Японские китобои знали о существовании редкой черной разновидности северных китов, имели для нее отдельное название «карасу» («ворон»), но не считали отдельным видом. Однако анализ митохондриальных геномов почти двух сотен китов Бэрда, в том числе и выделенных из музейных скелетов и китового мяса с японских рынков, позволил установить, что между черными китами и обычными серыми больше различий, чем между серыми и китами Арну. Это означает, что черные киты тоже должны рассматриваться как отдельный вид. Кстати, среди авторов статьи были и трое российских ученых — сотрудники биологического факультета МГУ имени М. В. Ломоносова И. Д. Федутин и О. А. Филатова и А. М. Бурдин из Камчатского филиала Тихоокеанского института географии ДВО РАН.
Автор благодарен фонду «Эволюция» за поддержку научно-популярных лекций, подготовка к которым помогла лучше осознать изложенный материал.
Работа выполнена при поддержке Российского научного фонда (проект 14-24-00155).
Литература
- Beddard F.E. A book of whales. N.Y., 1900.
- O’Leary M.A., Geisler J.H. The position of Cetacea within mammalia: phylogenetic analysis of morphological data from extinct and extant taxa // Syst. Biol. 1999. V.48. P.455–490.
- Arnason U., Gullberg A., Janke A. Mitogenomic analyses provide new insights into cetacean origin and evolution // Gene. 2004. V.333. P.27–34.
- Doi:10.1016/j.gene.2004.02.010.
- Graur D., Higgins D.G. Molecular evidence for the inclusion of cetaceans within the order Artiodactyla // Mol. Biol. Evol. 1994. V.11. P.357–364.
- Irwin D.M., Arnason U. Cytochrome b gene of marine mammals: phylogeny and evolution // J. Mammal. Evol. 1994. V.2. P.37–55.
- Doi:10.1007/BF01464349.
- Gatesy J., Hayashi C., Cronin M.A. et al. Evidence from milk casein genes that cetaceans are close relatives of hippopotamid artiodactyls // Mol. Biol. Evol. 1996. V.13. P.954–963.
- Gatesy J. More DNA support for a Cetacea/Hippopotamidae clade: the blood-clotting protein gene gamma-fibrinogen // Mol. Biol. Evol. 1997. V.14. P.537–543.
- Gatesy J., Milinkovitch M., Waddell V. et al. Stability of cladistic relationships between Cetacea and higher-level artiodactyl taxa // Syst. Biol. 1999. V.48. P.6–20.
- Kleineidam R.G., Pesole G., Breukelman H.J. et al. Inclusion of cetaceans within the order Artiodactyla based on phylogenetic analysis of pancreatic ribonuclease genes // J. Mol. Evol. 1999. V.48. P.360–368.
- Hasegawa M., Adachi J. Phylogenetic position of cetaceans relative to artiodactyls: reanalysis of mitochondrial and nuclear sequences // Mol. Biol. Evol. 1996. V.13. P.710–717.
- Montgelard C., Catzeflis F.M., Douzery E. Phylogenetic relationships of artiodactyls and cetaceans as deduced from the comparison of cytochrome b and 12S rRNA mitochondrial sequences // Mol. Biol. Evol. 1997. V.14. P.550–559.
- Ursing B.M., Arnason U. Analyses of mitochondrial genomes strongly support a hippopotamus-whale clade // Proc. Biol. Sci. 1998. V.265. P.2251–2255.
- Doi:10.1098/rspb.1998.0567.
- Gatesy J., Geisler J.H., Chang J. et al. A phylogenetic blueprint for a modern whale // Mol. Phylogenet. Evol. 2013. V.66. P.479–506.
- Doi:10.1016/j.ympev.2012.10.012.
- Shimamura M., Yasue H., Ohshima K. et al. Molecular evidence from retroposons that whales form a clade within even-toed ungulates // Nature. 1997. V.388. P.666–670.
- Doi:10.1038/41759.
- Shimamura M., Abe H., Nikaido M. et al. Genealogy of families of SINEs in cetaceans and artiodactyls: the presence of a huge superfamily of tRNA(Glu)-derived families of SINEs // Mol. Biol. Evol. 1999. V.16. P.1046–1060.
- Nikaido M., Rooney A.P., Okada N. Phylogenetic relationships among cetartiodactyls based on insertions of short and long interpersed elements: hippopotamuses are the closest extant relatives of whales // Proc. Natl. Acad. Sci. USA. 1999. V.96. P.10261–10266.
- Zhou X., Xu S., Yang Y. et al. Phylogenomic analyses and improved resolution of Cetartiodactyla // Mol. Phylogenet. Evol. 2011. V.61. P.255–264.
- Doi:10.1016/j.ympev.2011.02.009.
- Hassanin A., Delsuc F., Ropiquet A. et al. Pattern and timing of diversification of Cetartiodactyla (Mammalia, Laurasiatheria), as revealed by a comprehensive analysis of mitochondrial genomes // C. R. Biol. 2012. V.335. P.32–50.
- Doi:10.1016/j.crvi.2011.11.002.
- Thewissen J.G., Madar S.I. Ankle morphology of the earliest Cetaceans and its implications for the phylogenetic relations among ungulates // Syst. Biol. 1999. V.48. P.21–30.
- Thewissen J.G., Cooper L.N., Clementz M.T. et al. Whales originated from aquatic artiodactyls in the Eocene epoch of India // Nature. 2007. V.450. P.1190–1194.
- Doi:10.1038/nature06343.
- Thewissen J.G., Williams E.M., Roe L.J. et al. Skeletons of terrestrial cetaceans and the relationship of whales to artiodactyls // Nature. 2001. V.413. P.277–281.
- Doi:10.1038/35095005.
- Geisler J.H., Theodor J.M. Hippopotamus and whale phylogeny // Nature. 2009. V.458. E1–E4.
- Doi:10.1038/nature07776.
- Gingerich P.D., Haq M., Zalmout I.S. et al. Origin of whales from early artiodactyls: hands and feet of Eocene Protocetidae from Pakistan // Science. 2001. V.293. P.2239–2242.
- Doi:10.1126/science.1063902.
- McGowen M.R., Gatesy J., Wildman D.E. Molecular evolution tracks macroevolutionary transitions in Cetacea // Trends Ecol. Evol. 2014. V.29. P.336–346.
- Doi:10.1016/j.tree.2014.04.001.
- Zhou X., Sun F., Xu S. et al. Baiji genomes reveal low genetic variability and new insights into secondary aquatic adaptations // Nat. Commun. 2013. V.4. P.2708.
- Doi:10.1038/ncomms3708.
- Yim H.S., Cho Y.S., Guang X. et al. Minke whale genome and aquatic adaptation in cetaceans // Nat. Genet. 2014. V.46. P.88–92.
- Doi:10.1038/ng.2835.
- Kooyman G.L., Ponganis P.J. The physiological basis of diving to depth: birds and mammals // Annu. Rev. Physiol. 1998. V.60. P.19–32.
- Dolar M.L., Suarez P., Ponganis P.J. et al. Myoglobin in pelagic small cetaceans // J. Exp. Biol. 1999. V.202. P.227–236.
- Helbo S., Fago A. Functional properties of myoglobins from five whale species with different diving capacities // J. Exp. Biol. 2012. V.215. P.3403–3410.
- Doi:10.1242/jeb.073726.
- Noren S.R., Williams T.M. Body size and skeletal muscle myoglobin of cetaceans: adaptations for maximizing dive duration // Comp. Biochem. Physiol. A Mol. Integr. Physiol. 2000. V.126. P.181–191.
- Doi:10.1016/S1095-6433(00)00182-3.
- Dasmeh P., Kepp K.P. Bridging the gap between chemistry, physiology, and evolution: quantifying the functionality of sperm whale myoglobin mutants // Comp. Biochem. Physiol. A Mol. Integr. Physiol. 2012. V.161. P.9–17.
- Doi:10.1016/j.cbpa.2011.07.027.
- Holm J., Dasmeh P., Kepp K.P. Tracking evolution of myoglobin stability in cetaceans using experimentally calibrated computational methods that account for generic protein relaxation // Biochim. Biophys. Acta. 2016. V.1864. P.825–834.
- Doi:10.1016/j.bbapap.2016.04.004.
- Nery M.F., Arroyo J.I., Opazo J.C. Accelerated evolutionary rate of the myoglobin gene in long-diving whales // J. Mol. Evol. 2013. V.76. P.380–387.
- Doi:10.1007/s00239-013-9572-1.
- Mirceta S., Signore A.V., Burns J.M. et al. Evolution of mammalian diving capacity traced by myoglobin net surface charge // Science. 2013. V.340. P.1234192.
- Doi:10.1126/science.1234192.
- Schneuer M., Flachsbarth S., Czech-Damal N.U. et al. Neuroglobin of seals and whales: evidence for a divergent role in the diving brain // Neuroscience. 2012. V.223. P.35–44.
- Doi:10.1016/j.neuroscience.2012.07.052.
- McGowen M.R., Montgomery S.H., Clark C. et al. Phylogeny and adaptive evolution of the brain-development gene microcephalin (MCPH1) in cetaceans // BMC Evol. Biol. 2011. V.11. P.98.
- Doi:10.1186/1471-2148-11-98.
- McGowen M.R., Grossman L.I., Wildman D.E. Dolphin genome provides evidence for adaptive evolution of nervous system genes and a molecular rate slowdown // Proc. Biol. Sci. 2012. V.279. P.3643–3651.
- Doi:10.1098/rspb.2012.0869.
- Li Y., Liu Z., Shi P. et al. The hearing gene Prestin unites echolocating bats and whales // Curr. Biol. 2010. V.20. R55–R56.
- Doi:10.1016/j.cub.2009.11.042.
- Liu Y., Cotton J.A., Shen B. et al. Convergent sequence evolution between echolocating bats and dolphins // Curr. Biol. 2010. V.20. R53–R54.
- Doi:10.1016/j.cub.2009.11.058.
- Davies K.T., Cotton J.A., Kirwan J.D. et al. Parallel signatures of sequence evolution among hearing genes in echolocating mammals: an emerging model of genetic convergence // Heredity. 2012. V.108. P.480–489.
- Doi:10.1038/hdy.2011.119.
- Xu H., Liu Y., He G. et al. Adaptive evolution of tight junction protein claudin-14 in echolocating whales // Gene. 2013. V.530. P.208–214.
- Doi:10.1016/j.gene.2013.08.034.
- Shen Y.Y., Liang L., Li G.S. et al. Parallel evolution of auditory genes for echolocation in bats and toothed whales // PLoS Genet. 2012. V.8. e1002788.
- Doi:10.1371/journal.pgen.1002788.
- Liu Y., Rossiter S.J., Han X. et al. Cetaceans on a molecular fast track to ultrasonic hearing // Curr. Biol. 2010. V.20. P.1834–1839.
- Doi:10.1016/j.cub.2010.09.008.
- Sun Y.B., Zhou W.P., Liu H.Q. et al. Genome-wide scans for candidate genes involved in the aquatic adaptation of dolphins // Genome Biol. Evol. 2013. V.5. P.130–139.
- Doi:10.1093/gbe/evs123.
- McGowen M.R., Clark C., Gatesy J. The vestigial olfactory receptor subgenome of odontocete whales: phylogenetic congruence between gene-tree reconciliation and supermatrix methods // Syst. Biol. 2008. V.57. P.574–590.
- Doi:10.1080/10635150802304787.
- Feng P., Zheng J., Rossiter S.J. et al. Massive losses of taste receptor genes in toothed and baleen whales // Genome Biol. Evol. 2014. V.6. P.1254–1265.
- Doi:10.1093/gbe/evu095.
- Zhu K., Zhou X., Xu S. The loss of taste genes in cetaceans // BMC Evol. Biol. 2014. V.14. P.218.
- Doi:10.1186/s12862-014-0218-8.
- Kishida T., Thewissen J., Hayakawa T. et al. Aquatic adaptation and the evolution of smell and taste in whales // Zoological Lett. 2015. V.13. P.9.
- Doi:10.1186/s40851-014-0002-z.
- Fasick J.I., Robinson P.R. Mechanism of spectral tuning in the dolphin visual pigments // Biochemistry. 1998. V.37. P.433–438.
- Doi:10.1021/bi972500j.
- Fasick J.I., Robinson P.R. Spectral-tuning mechanisms of marine mammal rhodopsins and correlations with foraging depth // Vis. Neurosci. 2000. V.17. P.781–788.
- Dungan S.Z., Kosyakov A., Chang B.S. Spectral tuning of killer whale (Orcinus orca) rhodopsin: evidence for positive selection and functional adaptation in a cetacean visual pigment // Mol. Biol. Evol. 2016. V.33. P.323–336.
- Doi:10.1093/molbev/msv217.
- Newman L.A., Robinson P.R. Cone visual pigments of aquatic mammals // Vis. Neurosci. 2005. V.22. P.873–879.
- Doi:10.1017/S0952523805226159.
- Fasick J.I., Cronin T.W., Hunt D.M. et al. The visual pigments of the bottlenose dolphin (Tursiops truncatus) // Vis. Neurosci. 1998. V.15. P.643–651.
- Levenson D.H., Dizon A. Genetic evidence for the ancestral loss of short-wavelength-sensitive cone pigments in mysticete and odontocete cetaceans // Proc. Biol. Sci. 2003. V.270. P.673–679.
- Doi:10.1098/rspb.2002.2278.
- Meredith R.W., Gatesy J., Emerling C.A. et al. Rod monochromacy and the coevolution of cetacean retinal opsins // PLoS Genet. 2013. V.9. e1003432.
- Doi:10.1371/journal.pgen.1003432.
- Schweikert L.E., Fasick J.I., Grace M.S. Evolutionary loss of cone photoreception in balaenid whales reveals circuit stability in the mammalian retina // J. Comp. Neurol. 2016. V.524. P.2873–2885.
- Doi:10.1002/cne.23996.
- Nery M.F., González D.J., Opazo J.C. How to make a dolphin: molecular signature of positive selection in cetacean genome // PLoS One. 2013. V.8. e65491.
- Doi:10.1371/journal.pone.0065491.
- Wang Z., Yuan L., Rossiter S.J. et al. Adaptive evolution of 5’HoxD genes in the origin and diversification of the cetacean flipper // Mol. Biol. Evol. 2009. V.26. P.613–622.
- Doi:10.1093/molbev/msn282.
- Liang L., Shen Y.Y., Pan X.W. et al. Adaptive evolution of the Hox gene family for development in bats and dolphins // PLoS One. 2013. V.8. e65944.
- Doi:10.1371/journal.pone.0065944.
- Chen Z., Wang Z., Xu S. et al. Characterization of hairless (Hr) and FGF5 genes provides insights into the molecular basis of hair loss in cetaceans // BMC Evol. Biol. 2013. V.13. P.34.
- Doi:10.1186/1471-2148-13-34.
- Nery M.F., Arroyo J.I., Opazo J.C. Increased rate of hair keratin gene loss in the cetacean lineage // BMC Genomics. 2014. V.15. P.869.
- Doi:10.1186/1471-2164-15-869.
- Deméré T.A., McGowen M.R., Berta A. et al. Morphological and molecular evidence for a stepwise evolutionary transition from teeth to baleen in mysticete whales // Syst. Biol. 2008. V.57. P.15–37.
- Doi:10.1080/10635150701884632.
- São Pedro S.L., Alves J.M., Barreto A.S. et al. Evidence of positive selection of aquaporins genes from Pontoporia blainvillei during the evolutionary process of cetaceans // PLoS One. 2015. V.10. e0134516.
- Doi:10.1371/journal.pone.0134516.
- Wang J., Yu X., Hu B. et al. Physicochemical evolution and molecular adaptation of the cetacean osmoregulation-related gene UT-A2 and implications for functional studies // Sci. Rep. 2015. V.5. P.8795.
- Doi:10.1038/srep08795.
- Xu S., Yang Y., Zhou X. et al. Adaptive evolution of the osmoregulation-related genes in cetaceans during secondary aquatic adaptation // BMC Evol. Biol. 2013. V.13. P.189.
- Doi:10.1186/1471-2148-13-189.
- Tian R., Wang Z., Niu X. et al. Evolutionary genetics of hypoxia tolerance in cetaceans during diving // Genome Biol. Evol. 2016. V.8. P.827–839.
- Doi:10.1093/gbe/evw037.
- Foot N.J., Orgeig S., Donnellan S. Positive selection in the N-terminal extramembrane domain of lung surfactant protein C (SP-C) in marine mammals // J. Mol .Evol. 2007. V.65. P.12–22.
- Doi:10.1007/s00239-006-0083-1.
- Caballero S., Duchêne S., Garavito M.F. et al. Initial evidence for adaptive selection on the NADH subunit two of freshwater dolphins by analyses of mitochondrial genomes // PLoS One. 2015. V.10. e0123543.
- Doi:10.1371/journal.pone.0123543.
- McClellan D.A., Palfreyman E.J., Smith M.J. et al. Physicochemical evolution and molecular adaptation of the cetacean and artiodactyl cytochrome b proteins // Mol. Biol. Evol. 2005. V.22. P.437–455.
- Doi:10.1093/molbev/msi028.
- Bik E.M., Costello E.K., Switzer A.D. et al. Marine mammals harbor unique microbiotas shaped by and yet distinct from the sea // Nat. Commun. 2016. V.7. P.10516.
- Doi:10.1038/ncomms10516.
- Sanders J.G., Beichman A.C., Roman J. et al. Baleen whales host a unique gut microbiome with similarities to both carnivores and herbivores // Nat. Commun. 2015. V.6. P.8285.
- Doi:10.1038/ncomms9285.
- Shen T., Xu S., Wang X. et al. Adaptive evolution and functional constraint at TLR4 during the secondary aquatic adaptation and diversification of cetaceans // BMC Evol. Biol. 2012. V.12. P.39.
- Doi:10.1186/1471-2148-12-39.
- Shishido R., Ohishi K., Suzuki R. et al. Cetacean Toll-like receptor 4 and myeloid differentiation factor 2, and possible cetacean-specific responses against gram-negative bacteria // Comp. Immunol. Microbiol. Infect. Dis. 2010. V.33. e89–e98.
- Doi:10.1016/j.cimid.2010.03.003.
- Braun B.A., Marcovitz A., Camp J.G. et al. Mx1 and Mx2 key antiviral proteins are surprisingly lost in toothed whales // Proc. Natl. Acad. Sci. USA. 2015. V.30. P.8036–8040.
- Doi:10.1073/pnas.1501844112.
- Wada S., Oishi M., Yamada T.K. A newly discovered species of living baleen whale // Nature. 2003. V.426. P.278–281.
- Doi:10.1038/nature02103.
- Morin P.A., Baker C.S., Brewer R.S. et al. Genetic structure of the beaked whale genus Berardius in the North Pacific, with genetic evidence for a new species // Marine Mammal. Science. 2016.
- Doi:10.1111/mms.12345.
